Paper Digest: Cellen som et innebygd datasystem
Emily Reeves; 2. oktober 2023 Oversatt herfra
 Hvis livet bærer preg av en designers intensjon og fremsyn, er det fornuftig at ingeniørprinsipper vil være synlige på jobb i det designet. Gregory T. Reeves (ikke i slekt med forfatter) og Curtis E. Hriskchuk er en biolog-ingeniør-duo som forstår viktigheten av stedet der biologi og ingeniørkunst krysser hverandre. I en artikkel fra 2016 for Computational Biology Journal, "Survey of Engineering Models for Systems Biology," forklarer de hvordan ingeniørkonsepter kan brukes til å fremme systembiologi, som uten tvil er det mest designbaserte feltet innen biologi. Fra sammendraget:
Hvis livet bærer preg av en designers intensjon og fremsyn, er det fornuftig at ingeniørprinsipper vil være synlige på jobb i det designet. Gregory T. Reeves (ikke i slekt med forfatter) og Curtis E. Hriskchuk er en biolog-ingeniør-duo som forstår viktigheten av stedet der biologi og ingeniørkunst krysser hverandre. I en artikkel fra 2016 for Computational Biology Journal, "Survey of Engineering Models for Systems Biology," forklarer de hvordan ingeniørkonsepter kan brukes til å fremme systembiologi, som uten tvil er det mest designbaserte feltet innen biologi. Fra sammendraget:
I denne artikkelen identifiserer vi cellen som et innebygd datasystem og demonstrerer som sådan at systembiologi deler mange aspekter sammen med datasystemteknikk, elektroteknikk og kjemiteknikk.
Bilde 1. Celler
Likner mest systembiologi
Før de bringer designbaserte tilnærminger fra ingeniørfag til biologi, erkjenner forfatterne viktigheten av å identifisere ingeniør-(under)disiplinen som mest ligner på systembiologi. De spør:
 -Hvilken moderne datasystemarkitektur har den største likheten med cellens datasystem?
-Hvilken moderne datasystemarkitektur har den største likheten med cellens datasystem?
Generell databehandling, vitenskapelig eller høyytelses databehandling, distribuerte applikasjoner, DNA-databehandling eller innebygde systemer er alternativene i moderne datasystemarkitekturer. Ut av disse alternativene identifiserer de innebygde systemer som de mest lik det som skjer i en celle. For eksempel bemerker de at celler, lik innebygde datasystemer, inkluderer miljøinteraksjoner, samtidighet, reaktivitet, livlighet, motstandskraft og heterogenitet. De foreslår at modelleringstilnærmingene som brukes for innebygde systemer bør være svært nyttige for å utvikle systembiologi, som de definerer som "en systemteknisk tilnærming for å karakterisere biologiske prosesser."
-Vi viser at det cellulære innebygde systemparadigmet kan brukes til å konstruere prediktive modeller som er i stand til å sammenligne dynamiske (dvs. tidsbaserte) eksperimentelle resultater med modellprediksjoner og deretter avgrense hypoteser/spådommer i videre forskning.
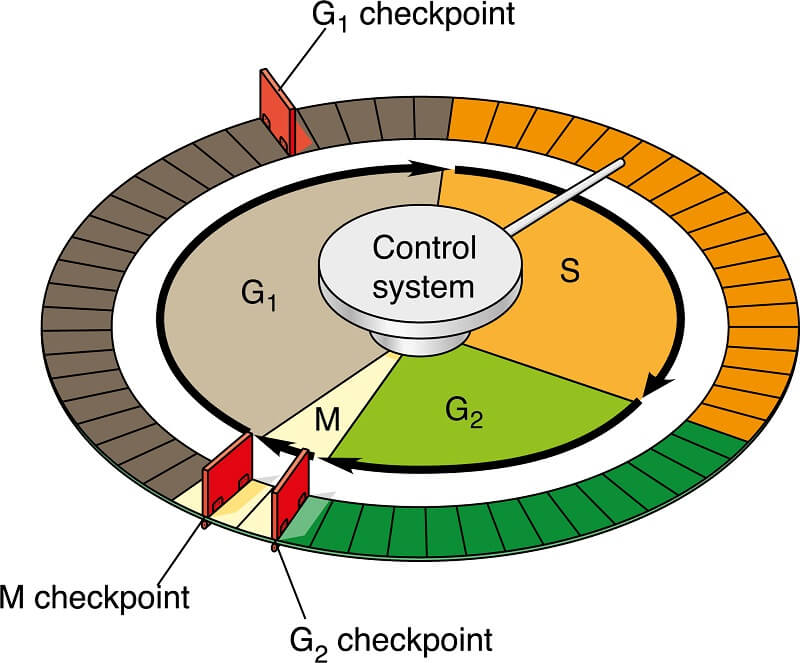
Bilde 2. Cellens regulatoriske syklus -via O. Grasso.
Tre modelleringsformalismer
 For å fremme vår forståelse av biologiske systemer, diskuterer de tre modelleringsformalismer: kontrollsystemmodellering, celleprosessmodellering og celleaktørmodellering. Dette er modelleringsteknikker som brukes i prosjektering for å designe og bygge komplekse systemer. Hvis organismer også er designet, som foreslått av ID-teorien, kan noen av disse verktøysettene fungere godt for en forbedret forståelse av levende systemer. La oss se på disse modelleringsformalismene litt mer detaljert.
For å fremme vår forståelse av biologiske systemer, diskuterer de tre modelleringsformalismer: kontrollsystemmodellering, celleprosessmodellering og celleaktørmodellering. Dette er modelleringsteknikker som brukes i prosjektering for å designe og bygge komplekse systemer. Hvis organismer også er designet, som foreslått av ID-teorien, kan noen av disse verktøysettene fungere godt for en forbedret forståelse av levende systemer. La oss se på disse modelleringsformalismene litt mer detaljert.
Kontrollsystemmodellering brukes til å beskrive kontrollsystemer som bruker tilbakemelding fra overvåkingssensorer for å holde utgangen innenfor et spesifikt område. Kontrollteori brukes i denne typen modellering, som studerer og forutsier forbigående og steady-state atferd i fysiske systemer, ved bruk av vanlige og differensial -ligninger. Prosessmodelleringen som de legger vekt på her, er opptatt av hva som må gjøres fremfor hvordan noe gjøres. I stedet for å fokusere utelukkende på en enzymatisk prosess for eksempel, er det ende-til-ende eller en sekvensiell algoritme i mange faser. Ifølge forfatterne er en av fordelene med prosessmodellering at den lar grunnleggende modeller integreres i større modeller, eller at høynivåmodeller kan få gradvis flere funksjoner lagt til etter behov. Denne modelleringsteknikken er ideell for biologiske veier og nettverk.
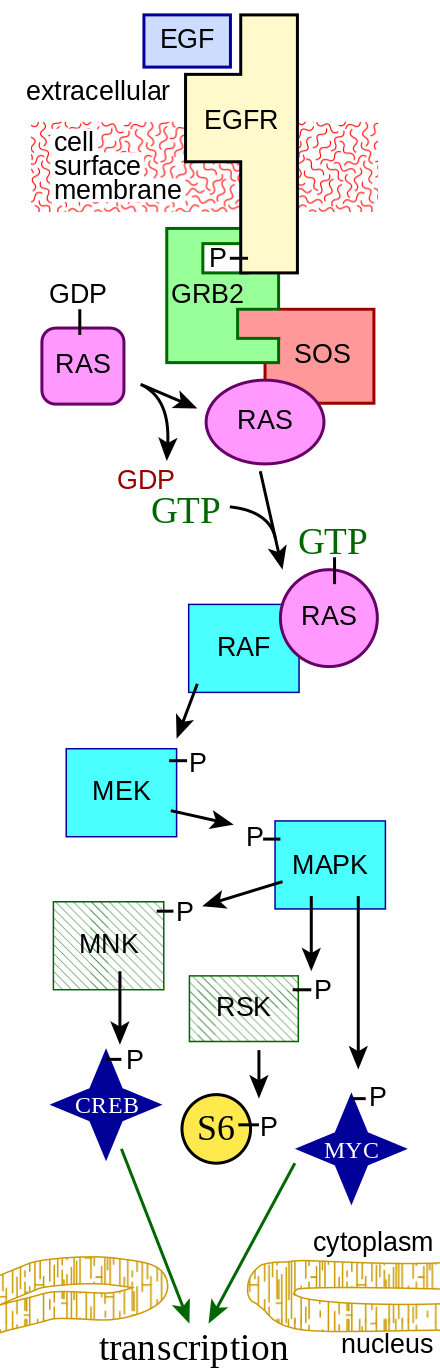
Bilde 3: ERK-stien-wikipedia-eng (biologi)
"Som allerede sett, passer denne formalismen godt inn i modelleringen av dataprosedyrer og programmer, men den kan også brukes direkte på biokjemiske veier og nettverk. For eksempel, i signaltransduksjon, er enzym/substrat-interaksjoner trukket sammen for å danne veier, som deretter kobles sammen for å danne nettverk. Faktisk har nyere arbeid brukt [SystemPlanlegging og Analyse] SPA-er for å modellere og analysere biokjemiske signalveier, for eksempel ERK-banen -lenke, som formidler mitogene og differensierings-signaler fra cellemembranen til kjernen.
Den tredje typen modell er aktør-modellering. Forfatterne anerkjenner denne typen modellering som ideelt egnet for å beskrive aktør-roboter i cellen, som ribosomet, lysosomet, etc. I disse tilfellene er det ingen sentralisert kontroll, men den globale atferden kommer fra den enkelte aktørens atferd -sml. blekksprut atferd -lenke . De gir som et eksempel en proteingradient som i seg selv er kontrollert av hastigheten på protein-syntese og protein-levetider.
Oppsummert
Denne artikkelen er en viktig illustrasjon på hvordan intelligent designteori kan brukes på et høyt nivå i akademiske omgivelser. Forfatterne argumenterer for viktigheten av ingeniørdisipliner (spesielt innebygde datasystemer) for biologi. De gir mange eksempler på lignende egenskaper mellom et innebygd datasystem og cellen. De minner leserne om å velge en modelltype som passer til hensikten med henvendelsen, og de håper at deres vurdering vil tjene som et utgangspunkt.

EMILY REEVES -bildet til venstre.
Emily Reeves er en biokjemiker, metabolsk ernæringsfysiolog og aspirerende systembiolog. Doktorgradsstudiene hennes ble fullført ved Texas A&M University i biokjemi og biofysikk. Emily er for tiden en aktiv kliniker for metabolsk ernæring og ernæringsgenomikk ved Nutriplexity. Hun liker å identifisere og designe ernæringsintervensjon for subtile medfødte metabolismefeil. Hun jobber også med stipendiater fra Discovery Institute og det større vitenskapelige samfunnet for å fremme integrering av ingeniørvitenskap og biologi. Hun tilbringer helgene sine på tur med mannen sin, brygger kombucha og løper i nærheten av Puget Sound.
Oversettelse via google oversetter og bilder ved Asbjørn E. Lund
 Hvis livet bærer preg av en designers intensjon og fremsyn, er det fornuftig at ingeniørprinsipper vil være synlige på jobb i det designet. Gregory T. Reeves (ikke i slekt med forfatter) og Curtis E. Hriskchuk er en biolog-ingeniør-duo som forstår viktigheten av stedet der biologi og ingeniørkunst krysser hverandre. I en artikkel fra 2016 for Computational Biology Journal, "Survey of Engineering Models for Systems Biology," forklarer de hvordan ingeniørkonsepter kan brukes til å fremme systembiologi, som uten tvil er det mest designbaserte feltet innen biologi. Fra sammendraget:
Hvis livet bærer preg av en designers intensjon og fremsyn, er det fornuftig at ingeniørprinsipper vil være synlige på jobb i det designet. Gregory T. Reeves (ikke i slekt med forfatter) og Curtis E. Hriskchuk er en biolog-ingeniør-duo som forstår viktigheten av stedet der biologi og ingeniørkunst krysser hverandre. I en artikkel fra 2016 for Computational Biology Journal, "Survey of Engineering Models for Systems Biology," forklarer de hvordan ingeniørkonsepter kan brukes til å fremme systembiologi, som uten tvil er det mest designbaserte feltet innen biologi. Fra sammendraget: -Hvilken moderne datasystemarkitektur har den største likheten med cellens datasystem?
-Hvilken moderne datasystemarkitektur har den største likheten med cellens datasystem? For å fremme vår forståelse av biologiske systemer, diskuterer de tre modelleringsformalismer: kontrollsystemmodellering, celleprosessmodellering og celleaktørmodellering. Dette er modelleringsteknikker som brukes i prosjektering for å designe og bygge komplekse systemer. Hvis organismer også er designet, som foreslått av ID-teorien, kan noen av disse verktøysettene fungere godt for en forbedret forståelse av levende systemer. La oss se på disse modelleringsformalismene litt mer detaljert.
For å fremme vår forståelse av biologiske systemer, diskuterer de tre modelleringsformalismer: kontrollsystemmodellering, celleprosessmodellering og celleaktørmodellering. Dette er modelleringsteknikker som brukes i prosjektering for å designe og bygge komplekse systemer. Hvis organismer også er designet, som foreslått av ID-teorien, kan noen av disse verktøysettene fungere godt for en forbedret forståelse av levende systemer. La oss se på disse modelleringsformalismene litt mer detaljert.